Détail de l'archive
Un modèle optimisé pour prédire la progression des espèces
16 janvier 2014 |
A l’heure de la mondialisation, les êtres humains et les marchandises ne sont pas les seuls à parcourir la planète dans tous les sens ; les plantes, les animaux et les micro-organismes peuvent atteindre (presque) n’importe quel endroit en un rien de temps et coloniser des espaces situés à des milliers de kilomètres de leur lieu d’origine. Pour pouvoir intervenir en temps et lieu voulus afin de lutter contre les invasions biologiques, il faut tout d’abord connaître la vitesse et les modalités de propagation des espèces.
Un modèle ancien mais une approche d’actualité
Les écologues utilisent depuis longtemps un modèle mathématique développé en 1937 par le biologiste Ronald Aylmer Fisher et le mathématicien Andreï Nikolaïevitch Kolmogorov pour prédire la vitesse moyenne de propagation des espèces. Il est basé sur les équations de diffusion-réaction qui sont également utilisées en physique, en chimie ou en cancérologie et décrit la progression des fronts d’invasion en assimilant les mouvements des individus à la diffusion et leur reproduction à la réaction. Pour un milieu donné, seuls deux paramètres simples à déterminer sont nécessaires au calcul : le coefficient de diffusion et le taux de croissance de l’espèce étudiée.
Malgré des atouts évidents, le modèle écologique présentait toutefois deux failles majeures : il n’avait jamais été testé en conditions contrôlées et il était incapable de reproduire la variabilité de la vitesse de propagation qui était visible dans la réalité. Avec leurs équipes, le biologiste Florian Altermatt de l’Eawag et le physicien Andrea Giometto de l’EPFL ont apporté de nouveaux éléments au modèle et vérifié les prédictions obtenues à l’aide d’essais de laboratoire.
Une invasion biologique dans un système en plexiglas
Les processus démographiques au sein des populations sont sujets à des variations qui ne dépendent pas uniquement des facteurs écologiques ; par exemple, différents individus ne produisent pas le même nombre de descendants même s’ils vivent dans les mêmes conditions. Les scientifiques ont décrit ce phénomène à l’aide de calculs de probabilités et ont ensuite complété le modèle d’une fonction permettant de quantifier les différences aléatoires et individuelles au niveau de la reproduction. Cette amélioration permet alors de déterminer la variabilité de la vitesse de propagation des espèces.
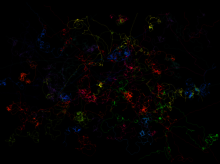
Les chercheurs ont ensuite comparé les calculs du modèle avec des observations en laboratoire. Pour leurs essais, ils ont rempli un tube de plexiglas de deux mètres de long de solution nutritive et ont introduit leur organisme d’étude, un cilié unicellulaire, à l’une de ses extrémités. Les déplacements individuels des ciliés ont été enregistrés par des caméras (voir photo), ce qui a permis de calculer les coefficients de diffusion. La vitesse de propagation en direction de la partie non encore colonisée du tube a été déterminée en se référant à une valeur critique de la densité de cellules dans le liquide.
Une forte corrélation a été observée entre les résultats des essais de laboratoire et les prédictions du modèle. Les chercheurs espèrent maintenant que la validation expérimentale et l’amélioration du modèle permettront une évaluation plus rapide et plus fiable de la colonisation et de sa dynamique dans les environnements naturels. Leur étude est paru le 7 Janvier 2014 dans la célèbre revue PNAS (Proceedings of the National Academy of Sciences).
Améliorer les chances de survie des espèces menacées
A l’opposé de sa lutte contre les invasions biologiques, l’homme met en œuvre toute une série de mesures pour protéger les espèces menacées et favoriser la réintroduction d’espèces animales ou végétales localement disparues. En 1988, des scientifiques américains se sont servis du modèle de Fisher-Kolmogorov pour étudier la population de la loutre de mer californienne sur le littoral pacifique. Cet animal avait été décimé par la chasse et, au début du XXe siècle, ses effectifs n’étaient plus que d’une centaine d’individus concentrés sur un seul site. Grâce à une réglementation très sévère, la population a pu se régénérer et les loutres se sont remises à coloniser les côtes vers le nord et vers le sud. Le modèle a permis de calculer la vitesse d’expansion de l’espèce et de prédire la recolonisation de ses anciens habitats. Andrea Giometto et les autres chercheurs espèrent maintenant que, dans sa nouvelle version, l’outil mathématique fournira également une indication de la variabilité de ce phénomène et permettra donc d’optimiser les mesures de protection.
Les déplacements des ciliés (Tetrahymena sp.) enregistrés pendant une minute sur une distance de quelques millimètres. Chaque tracé de couleur correspond à la trajectoire d’un individu. L’étude des patrons locaux de déplacement et de reproduction des ciliés permet de prédire la dynamique d’invasion sur plusieurs générations. © Eawag.
La caméra dont est équipé le microscope (au fond) permet d’enregistrer les mouvements des individus ; au premier plan, le tube de plexiglas qui a permis aux chercheurs de déterminer la vitesse de propagation des ciliés. © Eawag
Informations complémentaires
Florian Altermatt: +41 58 765 5592 (allemand et anglais); florian.altermatt@cluttereawag.ch
Andrea Giometto: +41 21 69 38027 (français, italien et anglais); andrea.giometto@clutterepfl.ch http://echo.epfl.ch
Article original
Giometto A, Rinaldo A, Carrara F & Altermatt F. Emerging predictable features of replicated biological invasion fronts. Proceedings of the National Academy of Sciences, DOI:10.1073/pnas.1321167110.
http://www.pnas.org/content/111/1/297.full.pdf+html