Abteilung Umweltmikrobiologie
Spezialisierter mikrobieller Metabolismus
Forschungsgebiete
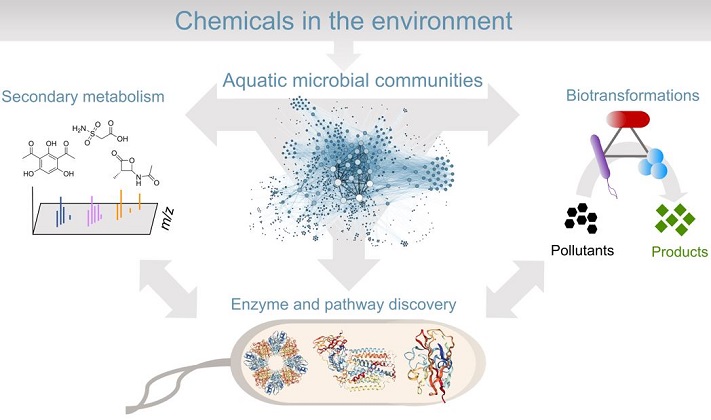
Mikroben kodieren eine bemerkenswerte Vielfalt von Enzymen, um chemische Substanzen in der Umwelt zu produzieren und abzubauen. Im Gegensatz zum Primärstoffwechsel bezeichnen wir hier als "spezialisierten Metabolismus" Stoffwechselwege mit eng regulierten Enzymen. Diese Enzyme werden oft von Genclustern für die Synthese, die Transformation und den Abbau bioaktiver Verbindungen kodiert. Dank neuen Sequenzierungsmethoden sehen wir heute, dass wir die funktionelle Vielfalt der spezialisierten Enzyme und Metaboliten, die von der Mehrheit der nicht kultivierten Mikroorganismen produziert werden, nur an der Oberfläche angekratzt haben. In unserer Gruppe versuchen wir, die folgenden Forschungsfragen zu beantworten:
- Welche Auswirkungen haben anthropogene Einflüsse auf den spezialisierten Metabolismus aquatischer Mikroben in Süsswasserökosystemen?
- Wie beeinflussen spezialisierte Stoffwechselprodukte die biologischen Abbauraten in mikrobiellen Konsortien?
- Können wir maschinelles Lernen nutzen, um die Funktion und Substratspezifität spezialisierter mikrobieller Enzyme, die am Schadstoffabbau beteiligt sind, vorherzusagen?
Bei unserem derzeitigen Schwerpunkt auf der letztgenannten Frage verwenden wir heterologe Expression in genetischen Modellsystemen und In-vivo- und In-vitro-Assays, um neue Enzyme und Stoffwechselwege aus nicht kultivierten Mikroben zu entdecken. Anschliessend nutzen wir unsere experimentellen Ergebnisse, um Modelle des maschinellen Lernens zu trainieren, um Muster der Substratspezifität und Funktion von Enzymen zu erlernen, die an der Biosynthese von Sekundärmetaboliten und dem biologischen Abbau von Schadstoffen beteiligt sind. Langfristig sind wir daran interessiert, Enzyme als Biokatalysatoren für umweltbiotechnologische Anwendungen wie die Verbesserung der Wasserqualität zu charakterisieren und zu entwickeln. Auf einer grundlegenderen Ebene sind wir auch an der ökologischen Rolle spezialisierter Enzyme und Metaboliten in mikrobiellen Gemeinschaften interessiert.
Für eine vollständige Publikationsliste siehe: Google Scholar
Ausgewählte Publikationen
- Marti, T.D., Schärer M.R., Robinson, S.L. (2023) Microbial Biocatalysis within Us: The Underexplored Xenobiotic Biotranformation Potential of the Urinary Tract Microbiota. CHIMIA. doi: 10.2533/chimia.2023.424.
- Robinson, S.L.*, Piel, J., & Sunagawa, S. A. (2021) A roadmap for metagenomic enzyme discovery. Natural Product Reports. doi: 10.1039/D1NP00006C.
- Robinson, S.L., Biernath, T., Rosenthal, C., Young, D., Wackett, L.P., & Martinez-Vaz, B.M. (2021) Development of the organonitrogen biodegradation database: teaching bioinformatics and collaborative skills to undergraduates Journal of Biology & Microbiology Education. 22(1), ev22i1.2351. doi: 10.1128/jmbe.v22i1.2351.
- Wackett, L.P. & Robinson, S.L. (2020) The ever-expanding limits of enzyme catalysis and biodegradation: polyaromatic, polychlorinated, polyfluorinated, and polymeric compounds. Biochemical Journal. 477(15), 2875–2891. doi: 10.1042/BCJ20190720.
- Robinson, S.L., Smith, M.D., Richman, J.E., Aukema, K.G., & Wackett, L.P. (2020) Machine learning-based prediction of activity and substrate scope for OleA enzymes in the thiolase superfamily. Synthetic Biology. 5(1), ysaa004. doi: 10.1093/synbio/ysaa004.