Abteilung Umwelttoxikologie
Entwicklung eines Proteinmarker-Panels zur Überwachung chemisch induzierter molekularer Reaktionen und des Schadensverlaufs in Fischzellen, um eine tierfreie Vorhersage der chemischen Toxizität zu ermöglichen
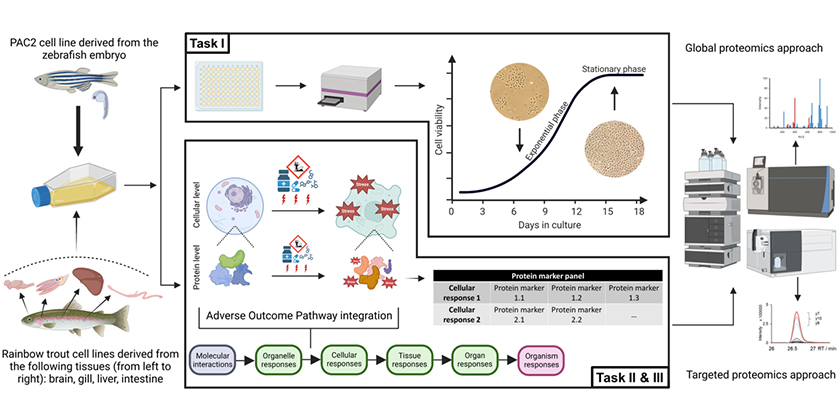
Für die Umweltrisikobewertung werden häufig Daten zur aquatischen Toxizität in Fischen benötigt. Gegenwärtig werden diese Daten immer noch hauptsächlich durch In-vivo-Tests an Fischen gewonnen. Diese Tests sind teuer und erfordern eine grosse Anzahl von Tieren, was sowohl wirtschaftliche als auch ethische Bedenken aufwirft. Da sich diese Tests zudem auf integrative Endpunkte wie Tod oder Wachstum stützen, liefern die erzielten Ergebnisse wenig mechanistische Informationen, was ihre Nützlichkeit für die Extrapolation auf nicht getestete Chemikalien oder auf andere Arten stark einschränkt. Alternative Toxizitätstests, bei denen permanente Fischzelllinien (in vitro) als Vorhersager für die Reaktionen von Fischorganismen auf eine chemische Exposition in vivo verwendet werden, bieten die Möglichkeit, einige dieser Bedenken auszuräumen und einen Ansatz zur Durchführung von aquatischen Toxizitätstests ohne Tiere zu bieten. Die derzeit existierenden Tests auf der Grundlage von Fischzelllinien funktionieren jedoch weitgehend als "Black Box", da sie sich auf eine eher kleine Anzahl von molekularen/zellulären Endpunkten stützen, und somit nur begrenzte Informationen über die Mechanismen der Toxizität liefern können.
Dieses PhD-Projekt zielt darauf ab, das Verständnis (i) der funktionellen Kapazitäten und der Gewebespezifität ausgewählter Fischzelllinien und (ii) ihrer molekularen Reaktionen, die bei chemischer Exposition ausgelöst werden, zu erweitern, wobei der Schwerpunkt auf der Proteomebene liegt. Es ist Teil eines grösseren Projekts, das darauf abzielt, ein modulares, rechnergestütztes Rahmenwerk zu entwickeln, das verschiedene Fischzelllinien und mechanistische Endpunkte integriert, um eine zweckmässige tierfreie Vorhersage der chemischen Toxizität für Fische zu ermöglichen. Das Projekt wird vom Schweizerischen Nationalen Forschungsprogramm 79 "Advancing 3Rs – Tiere, Forschung und Gesellschaft" finanziert.
Um die funktionelle Kapazität zu entschlüsseln und den Grad der Gewebespezifität, den die permanenten Fischzelllinien beibehalten, zu verstehen, wird eine auf Massenspektrometrie basierende globale Proteomanalyse durchgeführt. Dabei wird die Proteinexpression in den Zellen analysiert, die in den exponentiellen und stationären Wachstumsstadien des Zellkulturzyklus gesammelt wurden. Es werden mehrere Fischzelllinien von Zebrafischen und Regenbogenforellen analysiert. Proteomik- und Transkriptomikdaten (die von einem anderen Doktoranden im Rahmen des grosseren Projekts gesammelt wurden) werden dann integriert, um das Spektrum der molekularen Komponenten und Signalwege zu verstehen, die von jeder Zelllinie exprimiert werden, sowie um eine vergleichende Analyse für die verschiedenen Arten und Gewebe durchzuführen.
Um eine detaillierte Untersuchung der durch die chemische Exposition ausgelösten molekularen Reaktionen zu ermöglichen, wird ein Proteinmarker-Panel entwickelt. Dieses Panel umfasst Zebrafischproteine, die für bestimmte Toxizitätsmechanismen (wie z. B. oxidativen Stress) sowie für toxizitätsbedingte Auswirkungen, die in den Zellen während der Toxizität auftreten (wie z. B. Veränderungen des Energiestoffwechsels oder Umstrukturierungen des Zytoskeletts), charakteristisch sind. Gezielte Proteomik-Assays werden für Proteinmarker-Kandidaten mittels Selected Reaction Monitoring (SRM) etabliert. Im Vergleich zur globalen Proteomik ermöglicht diese Methode eine höhere Empfindlichkeit und einen schnelleren Probendurchsatz. Die Veränderungen in der Expression dieser Proteine werden dann in Zebrafisch-PAC2-Zellen gemessen unter Exposition auf gewisse Referenzchemikalien, von denen bekannt ist, dass sie spezifische Toxizitätsmechanismen auslösen. Ziel ist es, ein detailliertes, konzentrations- und zeitaufgelöstes Verständnis der molekularen Veränderungen zu erlangen, die dem zellulären Übergang von adaptiven zu adversen Reaktionen zugrunde liegen und zum Fortschreiten der Toxizität innerhalb der chemisch exponierten Zellen führen. Nach erfolgreicher Etablierung des ersten Proteinmarker-Panels werden die Untersuchungen auf weitere Mechanismen und Chemikalien sowie auf die vergleichende Erforschung von Proteinreaktionen in Zelllinien der Regenbogenforelle ausgeweitet. Die gewonnenen mechanistischen und quantitativen Daten werden auch in die jeweiligen Adverse Outcome Pathways (AOPs) integriert.






